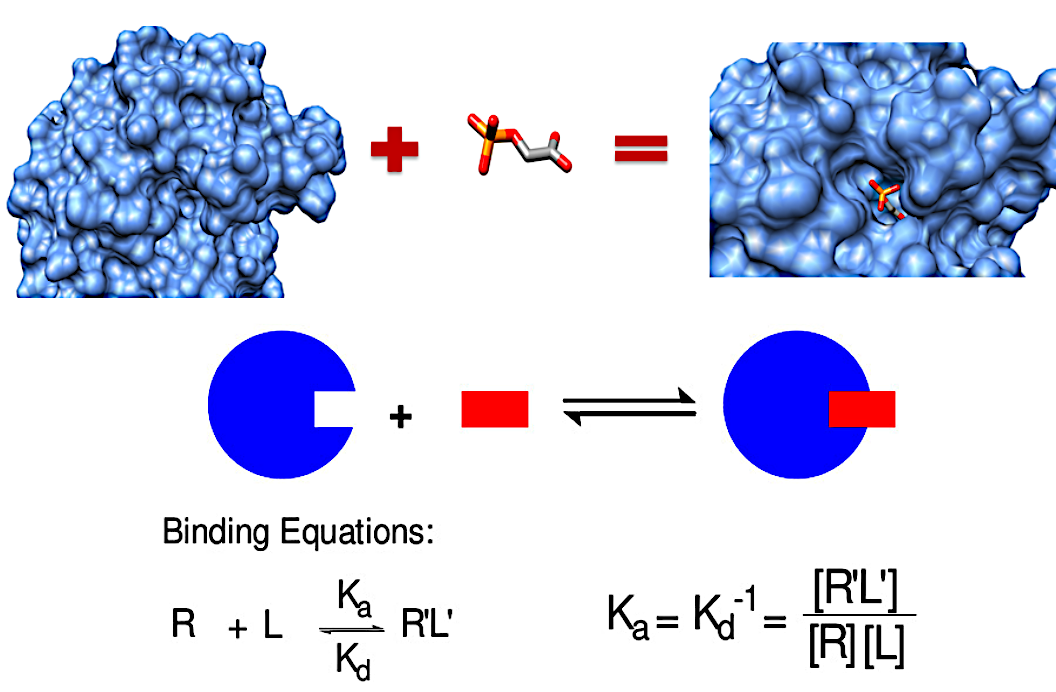
داکینگ مولکولی چیست؟
داکینگ مولکولی (Molecular Docking) یکی از روشهای مهم در زیستمحاسبات و شیمی محاسباتی است که برای پیشبینی نحوهی برهمکنش بین دو یا چند مولکول استفاده میشود.
در این روش، یک مولکول کوچک مانند لیگاند در برابر یک مولکول بزرگتر مانند پروتئین، DNA یا RNA قرار میگیرد تا بهترین حالت اتصال میان آنها شناسایی شود. نتیجهی نهایی، مدلی از کمپلکس پروتئین–لیگاند است که نشان میدهد لیگاند در کدام بخش از گیرنده قرار میگیرد و چگونه به آن متصل میشود.
هدف داکینگ مولکولی:
هدف اصلی از انجام داکینگ، یافتن بهترین حالت اتصال و محاسبهی انرژی اتصال (Binding Energy) بین مولکولها است.
با محاسبهی این انرژی، پژوهشگر میتواند:
پایداری اتصال بین مولکولها را ارزیابی کند
گروههای عاملی مؤثر در برهمکنش را شناسایی نماید
ترکیبات مؤثرتر را برای طراحی دارو انتخاب کند
اجزای اصلی برنامه های داکینگ مولکولی:
۱. الگوریتمهای جستوجو (Search Algorithms)
الگوریتمهای جستوجو وظیفه دارند لیگاند را در موقعیتها و زوایای مختلف نسبت به گیرنده قرار دهند تا همهی حالات ممکن بررسی شود. چند گروه مهم از این الگوریتمها عبارتند از:
روشهای سیستماتیک (Systematic Methods): جستوجوی گامبهگام تمام درجات آزادی مولکولها.
روشهای تصادفی (Stochastic Methods): شامل الگوریتمهای Monte Carlo، Tabu Search و الگوریتمهای ژنتیک برای تولید ساختارهای متنوع و انتخاب بهترین حالت بر اساس قوانین آماری.
روشهای شبیهسازی (Simulation Methods): بهرهگیری از دینامیک مولکولی و کمینهسازی انرژی برای یافتن ساختارهای پایدارتر.
۲. الگوریتمهای امتیازدهی (Scoring Functions)
پس از تولید حالتهای مختلف اتصال، الگوریتم امتیازدهی به هر ساختار نمرهای بر اساس انرژی محاسبهشده میدهد. رایجترین روشهای امتیازدهی شامل:
روشهای مبتنی بر دانش (Knowledge-based): استفاده از دادههای آماری حاصل از ساختارهای کریستالوگرافی پروتئین–لیگاند.
روشهای انرژیمحور (Energy Component Methods): در نظر گرفتن نیروهای الکترواستاتیکی، واندروالس، اثر حلال و تغییر شکل مولکولها.
روشهای تجربی (Empirical): تخمین انرژی پیوندی بر اساس دادههای تجربی.
امتیازدهی ترکیبی (Consensus Scoring): ترکیب چند روش برای افزایش دقت نهایی.
انواع داکینگ مولکولی:
داکینگها را میتوان بر اساس میزان انعطافپذیری مولکولها به دو نوع اصلی تقسیم کرد:
۱. داکینگ صلب (Rigid Docking)
در این نوع، هر دو مولکول بهصورت ثابت و بدون تغییر شکل در نظر گرفته میشوند. این مدل سریع است اما ممکن است با واقعیت زیستی تفاوت داشته باشد، زیرا پروتئینها در شرایط واقعی شکل خود را برای انطباق با لیگاند تغییر میدهند.
۲. داکینگ انعطافپذیر (Flexible Docking)
در این روش، حداقل یکی از مولکولها، معمولاً لیگاند، اجازه دارد تغییر شکل دهد تا بهترین حالت اتصال را بیابد. اگرچه این روش به محاسبات بیشتری نیاز دارد، اما دقت بسیار بالاتری نسبت به داکینگ صلب دارد.
کاربردهای داکینگ مولکولی در پژوهش و طراحی دارو:
داکینگ مولکولی کاربردهای گستردهای در علوم زیستی و داروسازی دارد، از جمله:
طراحی داروهای جدید: پیشبینی نحوهی اتصال ترکیبات شیمیایی به گیرندههای زیستی و انتخاب بهترین کاندیداها برای سنتز.
غربالگری مجازی (Virtual Screening): بررسی سریع هزاران ترکیب در پایگاههای داده مانند ZINC یا PubChem برای یافتن مولکولهای فعال.
درک مکانیسم اثر داروها: مشاهدهی نحوهی برهمکنش دارو با پروتئین هدف در سطح اتمی.
شناسایی جایگاههای فعال در پروتئینها: یافتن نواحی مناسب برای اتصال لیگاندهای جدید.
نرمافزارهایی مانند AutoDock، AutoDock Vina، PyRx، GOLD، DOCK و SwissDock از رایجترین ابزارهای مورد استفاده در این زمینه هستند.
چالشها و آیندهی داکینگ مولکولی:
اگرچه داکینگ ابزار بسیار مفیدی است، اما چالشهایی نیز دارد. بیشتر نرمافزارها پروتئین را بهصورت ثابت در نظر میگیرند و اثرات دینامیکی، حلال و یونها بهطور کامل لحاظ نمیشود. با این حال، پیشرفت روشهای دینامیک مولکولی (Molecular Dynamics)، یادگیری ماشین (Machine Learning) و هوش مصنوعی (AI) باعث شده پیشبینیها دقیقتر و نتایج قابل اعتمادتر شوند.
در آینده، ترکیب داکینگ با مدلهای یادگیری عمیق، مسیر طراحی داروهای هوشمند را متحول خواهد کرد.
در بخش ۲ از مجموعهی «مبانی داکینگ مولکولی» ، با الگوریتم های اصلی فرآیند داکینگ، یعنی الگوریتمهای جستوجو و توابع امتیازدهی، به طور دقیقتر آشنا خواهیم شد.
پیشنهاد ویژه
در مرکز پژوهشهای رایانهای ایلیا (ICRC) آموزش تخصصی نرمافزارهای AutoDock و AutoDock Vina، آمادهسازی ورودیها، تحلیل خروجیها و اجرای پروژههای داکینگ بهصورت کامل ارائه میشود.
آموزش مقدماتی مبانی داکینگ مولکولی
آموزش انجام داکینگ مولکولی با استفاده از نرم افزار آتوداک
آموزش نرم افزار آتوداک وینا
آموزش نرم افزار پایرکس
برای پژوهشگرانی که به دنبال یک محیط آماده برای اجرای این نرم افزارها هستند، فلش تخصصی ICRC Bio USB حاوی سیستم عامل لینوکس و نرم افزارهای مورد نیاز از جمله AutoDock می باشد.
https://icrcenter.ir/%D8%B5%D9%81%D8%AD%D9%87-%D8%AC%D8%AF%DB%8C%D8%AF/#wz-section-wzs110
این مرکز همچنین با ارائه سرورهای پرسرعت، امکان اجرای پروژه های سنگین محاسباتی را فراهم نموده است.
https://icrcenter.ir/services/#wz-section-wzs86
مرکز پژوهشهای رایانهای ایلیا — همراه شما در مسیر پژوهش و نوآوری
